Antecedentes
El gen del RNA ribosomal o la secuencia codificante del RNA ribosomal “rRNA” del 16S de ~ 1,550 pb, se encuentra en todos los organismos procariontes (bacterias y arqueobacterias), es una secuencia de DNA ampliamente utilizada en estudios filogénicos y una herramienta muy poderosa para la identificación taxonómica.
El gen 16S rRNA tiene varias regiones conservadas que son comunes a un gran número de especies bacterianas, y regiones variables, que son compartidas por menos especies. Es posible amplificar por PCR (reacción de polimerización en cadena) las regiones conservadas para una posterior identificación por secuenciación y su análisis, sin embargo, amplificar la región utilizando los cebadores universales no puede apuntar a todas las bacterias o arqueobacterias.
Utilizando la misma técnica del PCR para amplificar el gen completo 16S rRNA, secuenciando los productos y analizándolos contra las bases de datos del gen 16S, es posible identificar el género y en algunos casos la especie del microorganismo, dependiendo de la calidad de los datos ingresados en estas bases de datos disponibles, sin embargo, la identificación de los microorganismos utilizando para el análisis el gen 16S completo, es mucho más certero que analizando solo la región que se genera con los cebadores universales.
El objetivo de este servicio es que por medio de la amplificación de la mayor parte del gen 16S, con su posterior secuenciación y análisis, sea posible identificar el género de un microorganismo y tratar de identificar la especie con el mayor grado de certeza posible, considerando que esta identificación depende de la información y la calidad de las secuencias con las que cuentan las bases de datos de gen 16S rRNA.
También es muy importante considerar que para este tipo de identificación se deberá partir de un cultivo puro o una colonia única.
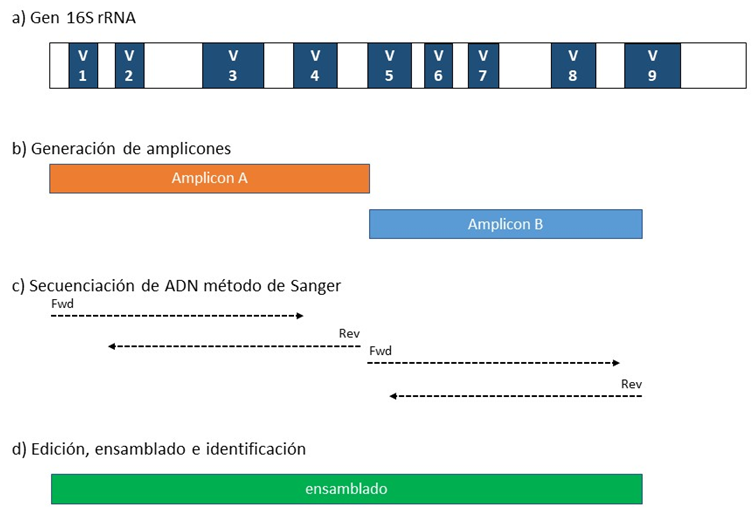
Figura 1. Secuenciación del gen 16S rRNA. a)Representación esquemática del gen 16S rRNA que muestra regiones variables (V1– V9) y regiones con secuencia conservadas (segmentos blancos). b)Segmento entre los nucleótidos 8 al 1,511 donde las secuencias se superponen, sirven como un sitio de unión de cebador para los amplicones de PCR "A" y "B". c)Secuenciación bidireccional de los productos de PCR "A" y "B" por el método de Sanger. d)Análisis de la secuencia ensamblada para identificar género y especie.
Descripción del Servicio
Utilizando la técnica del PCR para amplificar el gen completo 16S rRNA, con su posterior secuenciación por el método de SANGER y análisis, es posible identificar el género del microorganismo y tratar de identificar la especie con el mayor grado de certeza posible, esto último considerando que la identificación depende de la información y la calidad de las secuencias con las que cuentan las bases de datos disponibles del gen 16S rRNA.
Flujo de Trabajo
Entrega de Resultados
- Toda la información será transferida al cliente de forma segura y confidencial vía electrónica o digital.
- Los datos que serán entregados incluyen:
- Reporte de Resultados de los análisis solicitados.
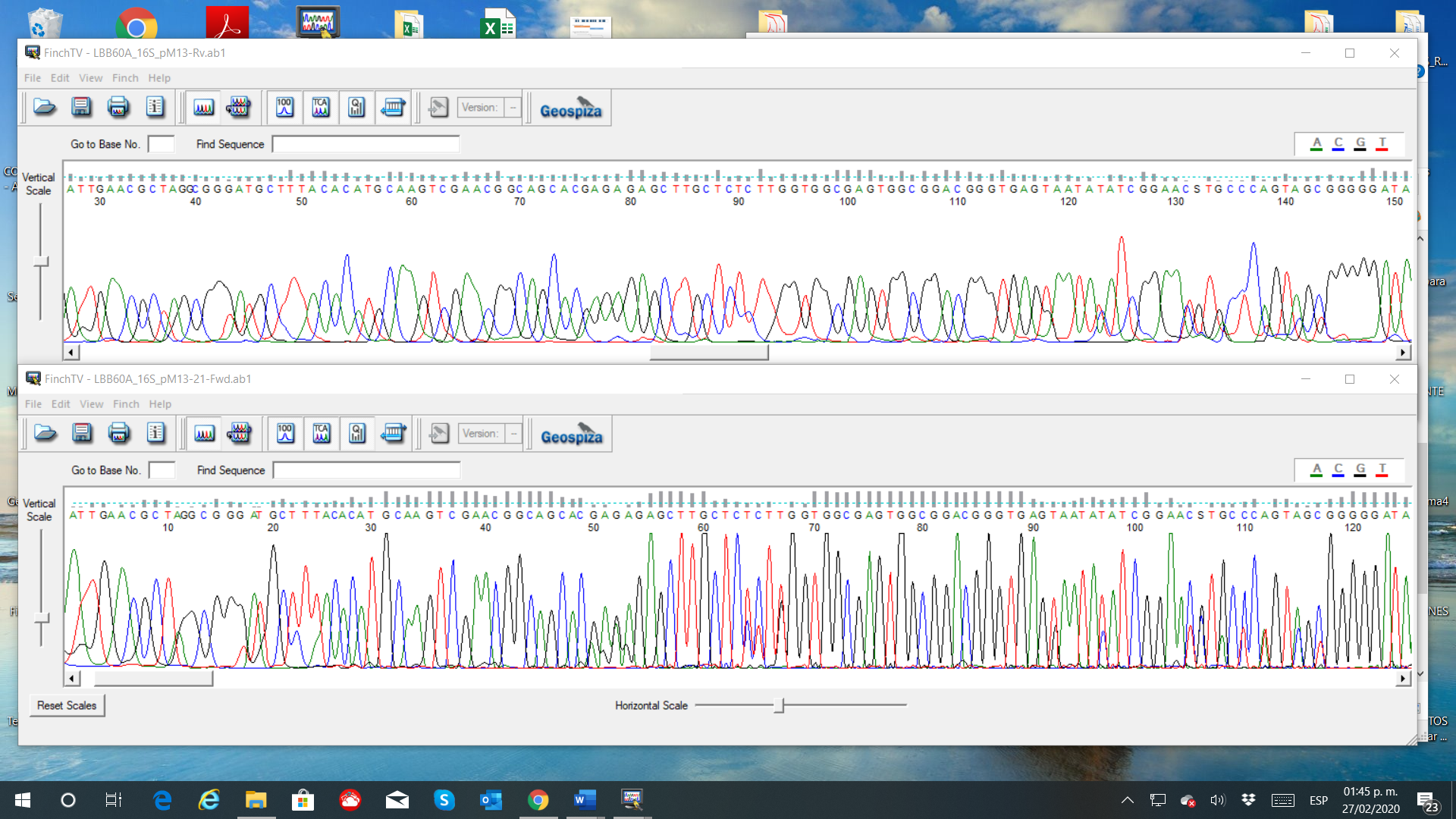
- Electroferogramas editados Forward y Revers de cada una de las muestras en formato.ab1.
- Archivos FASTA generados a partir de los electroferogramas.
- Archivo de ensamblado en formato txt por muestra.
- Es importante mencionar que, para la identificación, es posible lograr identificar la especie, pero esto dependerá de la información contenida en las bases de datos.
- El usuario tiene 3 meses posteriores a la entrega del reporte para recoger sus muestras y descargar sus datos, el laboratorio no se hace responsable de almacenarlos después de ese tiempo.
Los resultados serán entregados por el área de bioinformática a cargo de:
Archivos Entregables
| Archivo | Descripción |
| reporte_resultados.pdf | Identificación a nivel género y *especie. |
| archivo.AB1 | Secuencia de dato en formato de electroferograma. |
| archivo.fasta | Secuencia de dato editado en formato fasta. |
| ensamblado.txt | Secuencia de dato ensamblado en formato txt |
Ejemplo de Identificación
| Nombre de la Muestra | Descripción | % de Cobertura | % de Identidad | Longitud |
| Muestra | Alcaligenes faecalis | 99.00 % | 97,38% | 1,498nt |
Electroferograma
Requerimientos de la Muestra
- Partiendo de DNA genómico.
- Las muestras de DNA genómico deberán de ser entregadas en tubos eppendorf a una concentración de 50 ng/ul en 10 ul, en agua libre de nucleasas, no usar agua de DEPC a una relación 260/230 y 260/280 de 1.8 a 2.0.
- No entregar menos de 300 ng totales del DNA genómico.
- Se requiere de una fotografía de la integridad de cada una de las muestras como la cantidad de material que se entrega al laboratorio de Servicios genómicos.
- Las muestras deben estar perfectamente rotuladas de una manera legible e indeleble, el ID proporcionado será utilizado para la entrega de resultados.
- El laboratorio de Servicios genómicos no se hace responsable del empleo de muestras degradadas.
- Por ningún motivo serán recibidos las muestras si el usuario no entrega la hoja del Servicio de Identificación de Bacterias previamente llena y con la firma del responsable del proyecto.
- Incluyendo servicio de extracción de DNA genómico
- Requerimientos de extracción de DNA total de Bacterias
Formatos de Solicitud
Contacto
 Flor María Xochitl Zamudio Hernández
Flor María Xochitl Zamudio Hernández flor.zamudio@cinvestav.mx
flor.zamudio@cinvestav.mx Tel. +52 (462) 166-3000 Ext(s). 3126, 3024
Tel. +52 (462) 166-3000 Ext(s). 3126, 3024