Antecedentes
La región espaciadora interna transcrita, ITS por sus siglas en inglés (Internal Transcribed Spacer), se encuentra entre los genes del 18S rRNA, 5.8S rRNA y 28S, de hongos y otros eucariontes, y tiene un alto grado de variación en su secuencia, la cual es ampliamente utilizada para analizar la diversidad genética e identificación taxonómica.
Los estudios taxonómicos de hongos y otros eucariontes a menudo se basan en el grupo de genes nucleares ribosómicos, que incluye el 18S o una subunidad pequeña (SSU), subunidad 5.8S y genes de 28S rRNA o subunidad grande (LSU). Se ha encontrado que las secuencias entre el 18S-5.8S (ITS1) y la secuencia entre el 5.8S-28S (ITS2) son los marcadores más adecuados para el análisis filogenético y taxonómicos de hongos y otros acariotes, debido a sus secuencias variables.
El uso de la PCR para amplificar la región ITS1 e ITS2 a través de un solo producto de PCR (es una herramienta de amplio rango para la identificación de hongos y otros eucariontes), se pueden identificar por análisis de secuencia de nucleótidos el producto de PCR, seguido de la comparación de esta secuencia con secuencias conocidas y almacenadas en una base de datos.
Una de las dificultades que se presentan es el uso de la base de datos para la identificación a nivel de especie, esto debido a la falta de depuración de las secuencias, a la ambigüedad que pueden presentar estas secuencias y a la baja calidad de las secuencias ingresadas en estas bases de datos.
El objetivo de este servicio es cubrir la región ITS1 e ITS2, es cual será utilizado para la identificación del género y especie, en algunos casos se podrá identificar la especie con cierto grado de certeza, considerando que esta identificación depende de la información y la calidad que cuentan las bases de datos de la región ITS1 e ITS2.
También es muy importante considerar que para este tipo de identificación se deberá partir de un individuo, cultivo puro o colonia única, lo que estamos intentando asegurar es la identificación de la especie con esta metodología..
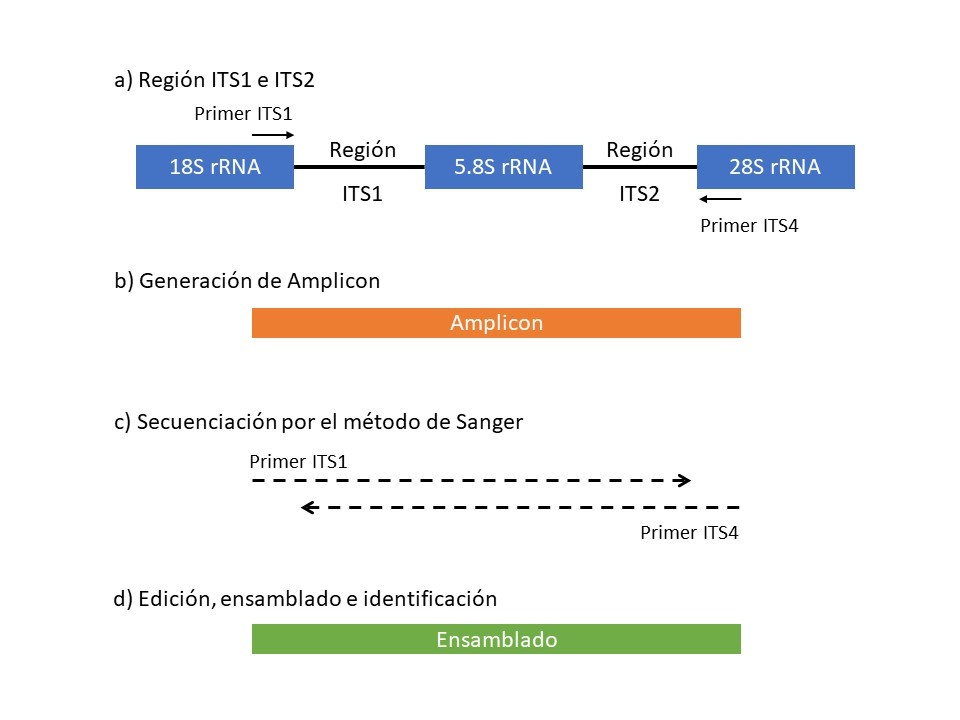
Figura 1. Secuenciación de la región ITS1 e ITS2.
(A) Representación esquemática del gen 18S rRNA, gen 5.8 rRNA y gen 28 rRNA, que muestra las regiones antigénicas ITS1 e ITS2 (B) Segmento de aproximadamente 650 nucleótidos del amplicon de PCR. (C) Secuenciación bidireccional del producto de PCR por el método de Sanger.(D) Análisis de la secuencia ensamblada para identificar género y especie.
Descripción del Servicio
Utilizando la técnica del PCR para amplificar la región ITS1 e ITS2, con su posterior secuenciación por el método de SANGER y análisis, es posible identificar el género del hongo y tratar de identificar la especie con el mayor grado de certeza posible, esto último considerando que la identificación depende de la información y la calidad de las secuencias con las que cuentan las bases de datos de la región ITS1 e ITS2 disponibles.
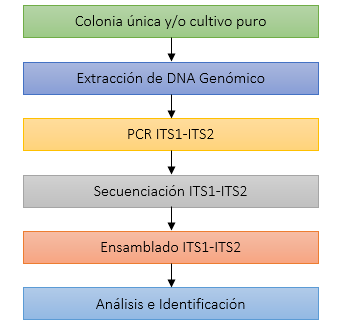
Flujo de Trabajo
Entrega de Resultados
- Toda la información será transferida al cliente de forma segura y confidencial vía electrónica o digital.
- Los datos que serán entregados incluyen:
- Reporte de Resultados de los análisis solicitados.
- Electroferogramas editados Forward y Revers de cada una de las muestras en formato.ab1
- Archivos FASTA generados a partir de los electroferogramas.
- Archivo de ensamblado en formato txt por muestra.
- Es importante mencionar que, para la identificación, es posible lograr identificar la especie, pero esto dependerá de la información contenida en las bases de datos.
- El usuario tiene 3 meses posteriores a la entrega del reporte para recoger sus muestras y descargar sus datos, el laboratorio no se hace responsable de almacenarlos después de ese tiempo.
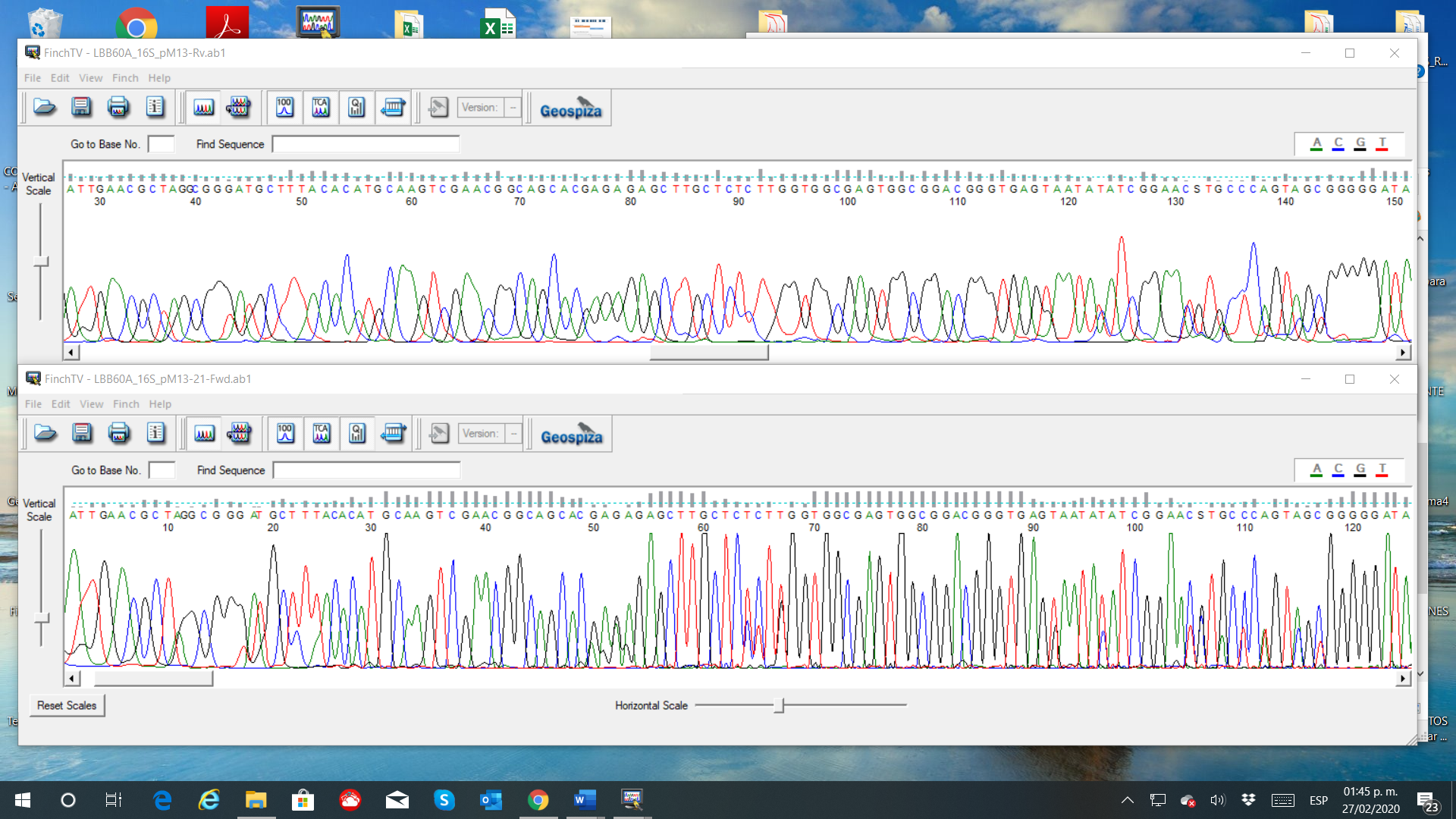
Electroferograma
Archivos Entregables
Identidad
| # | Nombre Muestra | Reference Description | Longitud | Similitud % | Sobreposición % |
| 1 | muestra | Trichoderma inhamatum | 528 | 99.43 | 100 |
Archivos entregables
| Archivo | Descripción |
| reporte_resultados.pdf | Identificación a nivel género y *especie. |
| archivo.AB1 | Secuencia de dato en formato de electroferograma. |
| archivo.fasta | Secuencia de dato editado en formato fasta. |
| ensamblado.txt | Secuencia de dato ensamblado en formato txt |
Los resultados serán entregados por el área de bioinformática a cargo de:
Requerimientos de la Muestra
- Partiendo de DNA genómico.
- Las muestras de DNA genómico deberán de ser entregadas en tubos eppendorf a una concentración de 50 ng/ul en 10 ul, en agua libre de nucleasas, no usar agua de DEPC a una relación 260/230 y 260/280 de 1.8 a 2.0.
- No entregar menos de 300 ng totales del DNA genómico.
- Se requiere de una fotografía de la integridad de cada una de las muestras como la cantidad de material que se entrega al laboratorio de Servicios genómicos.
- Las muestras deben estar perfectamente rotuladas de una manera legible e indeleble, el ID proporcionado será utilizado para la entrega de resultados.
- El laboratorio de Servicios genómicos no se hace responsable del empleo de muestras degradadas.
- Por ningún motivo serán recibidos las muestras si el usuario no entrega la hoja del Servicio de Identificación de Bacterias previamente llena y con la firma del responsable del proyecto.
- Incluyendo servicio de extracción de DNA genómico
- Requerimientos de extracción de DNA total de Hongos
Formatos de Solicitud
Contacto
 Flor María Xochitl Zamudio Hernández
Flor María Xochitl Zamudio Hernández flor.zamudio@cinvestav.mx
flor.zamudio@cinvestav.mx Tel. +52 (462) 166-3000 Ext(s). 3126, 3024
Tel. +52 (462) 166-3000 Ext(s). 3126, 3024